Agilent Microarray System |
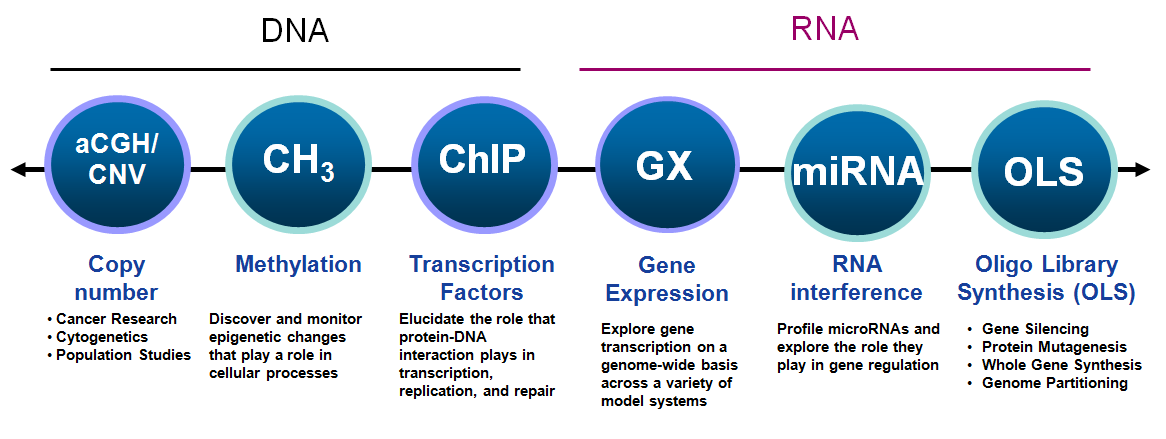
Agilent科技公司是一家多元化的高科技跨国公司,它于1999年从惠普公司分离出来,主要致力于通讯和生命科学两个领域内产品的研制开发、生 产销售和技术服务等工作。Agilent科技公司是分析仪器系统的领导供应商,其产品正在化学、环保、食品、医药和生命科学领域中广泛使用。 Agilent的微阵列解决方案基于一个集成的、灵活的开放式基因表达分析流程。每个部分的设计均可与我们系统中的其它部分或者客户已有的系统相兼容,以 加快客户的分析过程。 Agilent基因芯片特点 原位合成芯片-Agilent SurePrint Technology Agilent公司专利的喷点式原位合成技术(SurePrint技术),为您快速提供超高精准度(99%)长链(-60mer)载玻片式芯片。生产的60-mer寡核苷酸阵列比短寡核苷酸阵列检测灵敏度高八倍。
Agilent全自动进样、自动聚焦的双激光同步扫描仪,为您提供可靠的数据和谱图。Agilent 2100 生物分析仪已经取代了繁琐的凝胶电泳技术成为RNA 样品质量控制(QC) 的行业标准。同时在DNA 片段分析和蛋白样品SDS-PAGE 分析方面也正在迅速取代凝胶电泳技术。
Agilent 芯片种类齐全,应用广泛
Agilent 芯片规格
Genespring 强大的配套分析软件 Agilent-GeneSpring软件是目前应用最广泛的基因芯片数据分析软件,功能强大,界面友好,可以使您快速具备独立分析基因芯片数据的能力,从海量的芯片数据中轻松解脱出来。
Agilent芯片平台一览
技术特点1.独特的SurePrint原位合成技术 SurePrint技术依靠强大的探针设计运算法则和准确的确认方法,无需“光掩膜”,并利用工业规模的喷墨打印技术和独特的质控程序来合成芯片。正因为有了这项技术,安捷伦的芯片制造产业也得到蓬勃发展。 2.双色芯片检测 可以根据客户的需要,将相互比较的两个样本分别用不同的荧光标记,杂交在同一张芯片上。相较于到传统的单色芯片检测,此方法可以发现更微小的表达差异,并有效地控制实验的批次差异。 3.用量少 只需200ng 总RNA即可用来进行标记实验(不包括质检所消耗的量),大大降低了RNA抽提的难度,能让客户利用剩下的总RNA进行验证。 4.灵活方便的定制服务 安捷伦公司除了提供成品芯片外,还开放了一个免费的芯片设计平台,让客户可以根据自己的需求设计专有的芯片,同时亦可让客户分享其他客户的定制芯片,满足客户不同的研究需要。 检测原理
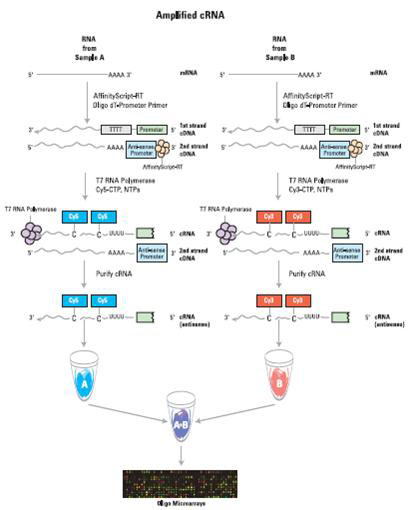
双色荧光标记表达谱芯片的基本原理就是,用两种不同的荧光分别标记实验组和对照组扩增后的cRNA,调整好两组的杂交用量,然后与表达谱芯片杂交,通过扫描芯片上两种荧光(红、绿)的相对强度比率,两组RNA有差异的部分会出现颜色偏移,从而计算出相应基因表达量是上调还是下调。如果选择做单色荧光标记的表达谱芯片检测,只需用一种荧光标记一个样本进行杂交即可。
样本要求来源于新鲜组织或者细胞 RNA浓度:≥100ng/μL,溶解在无核酸酶的水中 琼脂糖凝胶电泳显示无基因组DNA污染,无RNA降解 260/280:1.8-2.1 260/230:≥1.8 28S/18S:1.8-2.1 (不作硬性规定,如达不到,至少应满足在1.4-1.6之间) 检测服务流程
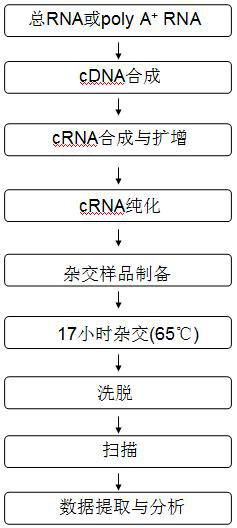
1.对待测样品进行NANODROP和2100 Bioanalyzer质检。 2.取200ng的总RNA或者poly A+ RNA进行cDNA合成 3.以上步合成的cDNA为模板进行cRNA转录合成。 4.cRNA纯化。 5.如果选择双色标记检测,用Cy-3和Cy-5对两组样本分别行进标记;如果选择单色标记检测,则用Cy-3对样本进行标记即可。然后对标记后的样本进行纯化、定量,检测标记是否达标。 6.如果是双色标记检测,将标记达标的两组RNA等量混合,同时与表达谱芯片杂交;如果是单色标记检测,只需取一定量的标记达标的RNA与表达谱芯片杂交。 7.杂交完毕后,对芯片洗脱。 8.芯片扫描 9.从扫描出来的.tiff文件上提取数据,并对数据归一化,各处初步的基因表达差异分析。 应用领域
1.疾病研究 研究疾病的基因表达时空特征,揭示疾病的分子基础 研究疾病的基因表达差异,寻找疾病分型的分子指标 病治疗前后的基因表达差异研究,从基因组学上评估其时效性 2.临床诊断 中医学孔临床诊断的分子基础研究 3.药物筛选 药物药理机制分析,药物的毒性毒理研究 PUBLICATION:[1] Bolton JJ, et al. Aberrant Expression of Critical Genes during Secondary Cell Wall Biogenesis in a Cotton Mutant, Ligon Lintless-1 (Li-1). Comp Funct Genomics. 2009:659301. [2] Mokry M, et al. Accurate SNP and mutation detection by targeted custom microarray-based genomic enrichment of short-fragment sequencing libraries. Nucleic Acids Res. 2010 Feb 17. [3] Rothgiesser KM, et al. Acetylation of p65 at lysine 314 is important for late NF-k(kappa)B-dependent gene expression. BMC Genomics. 2010 Jan 11;11:22. [4] van Zutphen T, et al. Adaptation of Hansenula polymorpha to methanol: a transcriptome analysis. BMC Genomics. 2010 Jan 4;11:1. [5] Haugen AC, et al. Altered gene expression and DNA damage in peripheral blood cells from Friedreich's ataxia patients: cellular model of pathology. PLoS Genet. 2010 Jan 15;6(1):e1000812. [6] Okubo Y, et al. Alternative Route for Glyoxylate Consumption During Growth on Two-carbon Compounds by Methylobacterium extorquens AM1. J Bacteriol. 2010 Apr;192(7):1813-23. [7] Cazalet C, et al. Analysis of the Legionella longbeachae Genome and Transcriptome Uncovers Unique Strategies to Cause Legionnaires' Disease. PLoS Genet. 2010 Feb 19;6(2):e1000851. [8] Mikkonen L, et al. Androgen receptor and androgen-dependent gene expression in lung. Mol Cell Endocrinol. 2010 Apr 12;317(1-2):14-24. [9] Chandriani S, et al. Array-based transcript profiling and limiting-dilution RT-PCR analysis identify additional latent genes in KSHV. J Virol. 2010 Mar 10. [10] Kankare M, et al. Changes in gene expression linked with adult reproductive diapause in a northern malt fly species: a candidate gene microarray study. BMC Ecol. 2010 Feb 1;10:3.
|
||